進化系統樹について、素人がまとめました。高校生程度(物化・数III)の知識を想定しています。素人がまとめたので嘘ばっかりかと思います。 間違っているところあれば、 Twitter : homemaku までツッコミください。
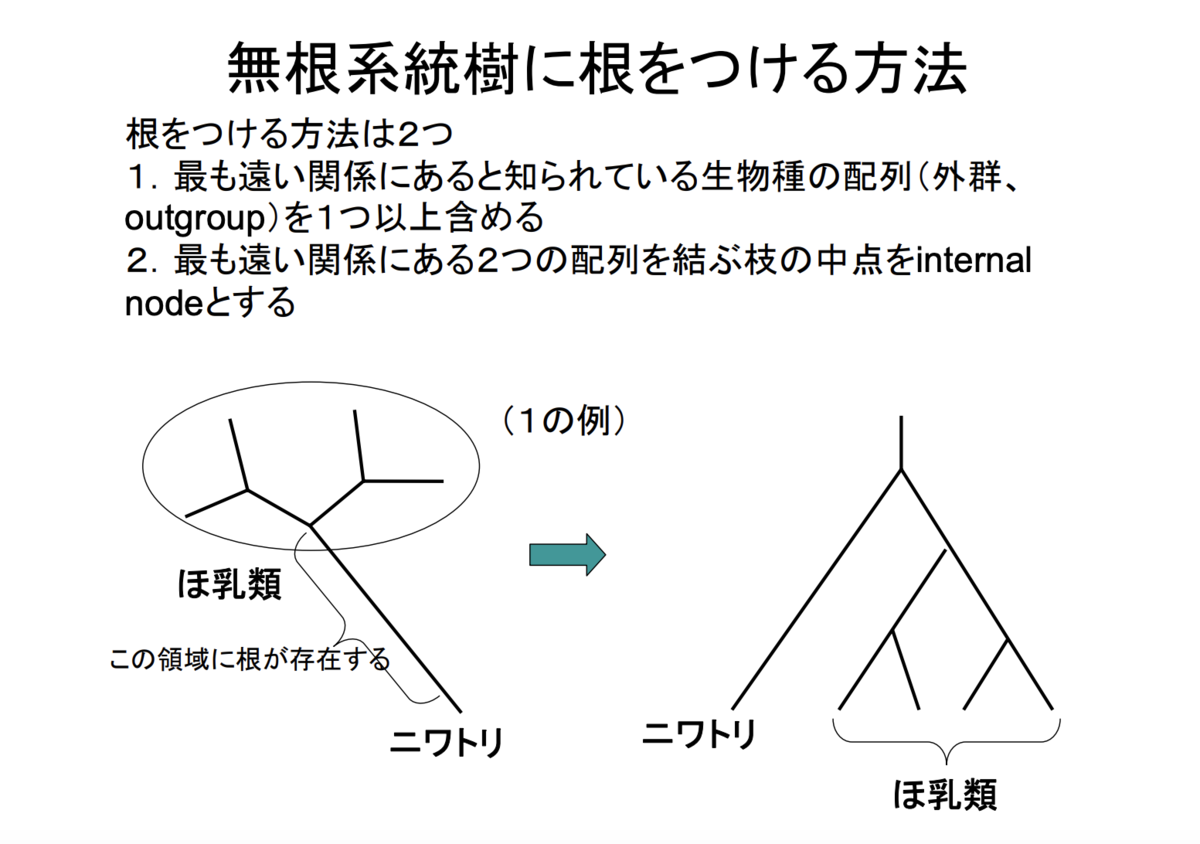
一種類の祖先ががあるやつは有根系統樹、祖先がいないものを系統樹としてグルーピングしたものは無根系統樹、となります。 でもまあ無根系統樹も、一番長い枝を真ん中で折ってrootにするとそれっぽくなります。

クラスタリング(グルーピング)に目が行きがちですが、有根系統樹も、無根系統樹も「枝の長さが大事」です。これが計算された遺伝的距離を表しています。
進化系統樹の作り方の流れは、大きく二段階に別れます。
距離行列の作成も、系統樹の作成も、どの方式を選ぶか、どの変異を選ぶかで、うまく作成できるかどうかが変わってきます。 だから生物横断のでかい系統樹ってなかなか作りにくいのかもしれません
距離行列はこんなやつです。
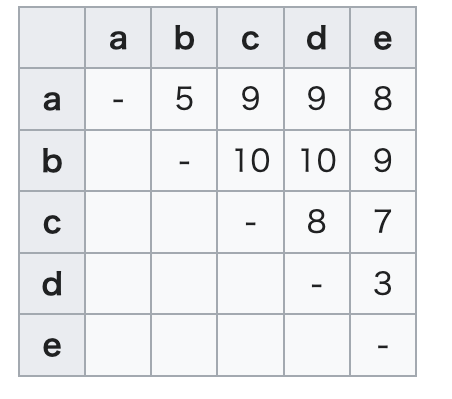
距離行列の作成方法はいろいろ有ります。 単純に、塩基の変異数を下記のように数えた例が距離行列として使われたりするそうです。
で大いに中身が変わってきそうです。
DNAの特定部分やタンパク質、トランスポゾンの転移数などに着目するSINE法など、方法はいろいろ有りそう。 また、変更と挿入、削除の重み付の違いや、同一タンパク質への翻訳となるが塩基上では変異となるものをどう扱うかなど、 チューニングがかなりある。
種間差によって、分類に最適な様々な方法が有りますが、なかなか全世代統一的に図るなどは難しい。 どの部位に着目するかで結果が大きく異なったりするようだ。
Wikipediaによれば下記がメジャーそうです。
UPGMA
近隣結合法
京都大学の阿久津先生の授業pdfが死ぬほど分かりやすいです。

上記の阿久津先生のプレゼンにより、Lengthの数値などは読めるようになりました。
NJ法は、距離Dについて下記式が成り立ちます。 種a, b間の分岐ノード(仮想的な共通祖先x)がいたとして、D(a,b)= D(a,x)+D(b,x)
今世紀起こったCOVID19の進化などを除けば、進化過程を見た人間はいなく、化石標本も十分でないため 系統樹の正当性の検証、すなわち、距離行列の方式選定と系統樹形状の正当性の検証は、 遺伝子の変異確率を使って、サイコロを振る(モンテカルロ法)再現実験で行っているそうです。
今メジャーなプロセスは下記のようになるそうです。
これは、信頼推定した区間の数値である。
68–95–99.7 rule のように、そのクラスタリングが何σに入るかを見る。例えば正規分布であれば、そのクラスタが2σに収まれば、95が表示される。 100は信頼推定したrootである。
ただ、塩基などの変異の確率を仮定してあるが(AATATがAATTTになる確率など) 反応場の問題もあるから、なかなか既知の値をよその種類から持ってくるのも難しそう。 ミトコンドリアは、直近1万年くらいをみるのに便利らしい。(ミトコンドリアは細胞質なのでオカンからやってくるから。その割に変異多いね)
@2355toby